BIONANOSENSOR DE NANOTUBO DE CARBONO COM FAGO ADSORVIDO PARA DETECÇÃO DE E.COLI
Capítulo de livro publicado no livro do I Congresso Latino-Americano de Segurança de Alimentos. Para acessa-lo clique aqui.
DOI: https://doi.org/10.53934/08082023-25
Este trabalho foi escrito por:
Delaine Meireles Gouvêa Boggione ; Felipe Ramalho Estanislau ; Regina Célia Santos Mendonça : Igor José Boggione Santos
Resumo: A ampla utilização de antibióticos e o subsequente surgimento de microrganismos que são resistentes a eles levou a um aumento do interesse em técnicas alternativas para controlar os patógenos e os deterioradores na indústria de alimentos e outros campos. Escherichia coli O157:H7 é um patógeno significativo que pode estar presente em alimentos e causar infecções intestinais graves em indivíduos. Uma possível alternativa para detectar esse patógeno em alimentos e eliminá-lo é através da utilização de bacteriófagos. Estes são vírus hospedeiro-específicos para e causam lise e morte celular. O objetivo desta pesquisa é empregar o bacteriófago UFV-AREG1 em nanotubos de carbono de paredes múltiplas (MWCNT) para estabelecer um nanosensor tensiométrico.
Palavras–chave: E. coli O157:H7, Bacteriófago, MWCNT, Nanobiosensor
Abstract: The widespread use of antibiotics and the subsequent emergence of microorganisms that are resistant to them has led to increased interest in alternative techniques for controlling pathogens and spoilage in the food industry and other fields. Escherichia coli O157:H7 is a significant pathogen that can be present in food and cause severe intestinal infections in individuals. A possible alternative to detecting this pathogen in food and eliminating it is through the use of bacteriophages. These are host-specific viruses for and cause lysis and cell death. The goal of this research is to employ the bacteriophage UFV-AREG1 on multi-walled carbon nanotubes (MWCNT) to establish a tensiometric nanosensor.
Keywords: E. coli O157:H7, Bacteriophage, MWCNT, Nanobiosensor
INTRODUÇÃO
Garantir a segurança alimentar para a população global é uma preocupação compartilhada por muitos países. Apesar da implementação de inúmeras medidas preventivas pelas agências reguladoras na última década, muitos indivíduos ainda sofrem de doenças veiculadas por alimentos (DTA) causadas por patógenos conhecidos, resultando em hospitalização ou até mesmo morte (1), (2).
Nos Estados Unidos (EUA), a Escherichia coli (O157:H7) é um dos principais culpados de doenças veiculadas por alimentos (3). Este patógeno é responsável pela muitos dos surtos de intoxicação alimentar. Os agentes antimicrobianos convencionais induzem uma resposta SOS na Escherichia coli Enterohemorrágica (EHEC), que desencadeia a liberação de uma poderosa toxina Shiga. Esta toxina é responsável por grande parte da gravidade e fatalidades associadas às infecções por EHEC. O gado serve como hospedeiro natural para EHEC, e os produtos pecuários são a fonte de aproximadamente 75% dos surtos de EHEC relacionados ao consumo de alimentos. De acordo com o Ministério, o consumo de carne bovina do Departamento de Agricultura dos EUA (USDA) atingiu em 2016 58,74 milhões de toneladas, com consumo estimado de 63 milhões Tonelagem em 2018 (4). Este alimento de origem animal altamente consumido estimula o desenvolvimento de formas mais eficazes de reduzir ou eliminar microrganismos patogênicos veiculados por esses alimentos (5).
O estudo dos patógenos causadores da DTA é realizado pela indústria alimentícia por meio de testes microbianos. Esses testes visam estabelecer os limites aceitáveis para esse patógeno e suas toxinas nos alimentos, bem como o prazo de validade e a segurança dos alimentos (Lopez-Velasco et al., 2015; HOORFAR, 2011). Portanto, é imperativo desenvolver um método rápido, e preciso para detectar bactérias patogênicas que causam DTA. Tem crescido o desenvolvimento e implementação de bionanossensores com baixos limites de detecção, incluindo bacteriófagos e sensores baseados em lecitina, bem como a utilização de nanomateriais e outras técnicas mais específicas (7).
A especificidade dos bacteriófagos para os procariontes-alvo, sua incapacidade de infectar células eucarióticas, sua capacidade de autorreplicação e sua ocorrência comum na natureza os tornam um candidato principal para agentes de biocontrole em alimentos (8,9). Os bacteriófagos também podem ser utilizados na criação de sensores, sendo sua imobilização natural utilizada para emitir um sinal que pode indicar a presença de determinadas moléculas. Ao imobilizar fagos em nanomateriais, o sinal indicativo pode ser intensificado (10).
Os nanotubos de carbono (CNTs) são um nanomaterial promissor para utilização em nanobiosensores, devido às suas características eletrônicas e capacidade de absorção de diversas substâncias (11,12). Esses CNTs já estão sendo empregados em interações de superfície com biomoléculas, como proteínas, peptídeos e nucleotídeos, bem como bacteriófagos para aumentar a eficácia como um produto terapêutico plausível (10,13).
O bacteriófago UFV-AREG1 foi isolado a partir de águas residuais de estábulo, mostrando especificidade para Escherichia coli O157: H7 (CDC EDL-933), E. coli 0111 (CDC O11ab) e E. coli (ATCC 23229). Bacteriófago UFV-AREG1 pode atingir concentrações acima de 1012 PFU/mL em condições de laboratório quando o hospedeiro é a E. coli O157: H7 (14).
Portanto, este projeto tem como objetivo imobilizar o bacteriófago UFV-AREG1 em nanotubos de carbono de paredes múltiplas (MWCNT) para a construção de um nanobiosensor para detecção de E. coli O157:H7.
2. MATERIAL E MÉTODOS
2.1 Material e micro-organismos
O fago será UFV- AREG1 específico para E. coli O157:H7, do Laboratório de Microbiologia de Patógenos de Origem Alimentar e Hídrica (LAMPOAH) da Universidade Federal de Viçosa (UFV). O nanotubo de carbono (MWCNTs, chamado aqui apenas de CNT) de paredes múltiplas serão obtidos do Laboratório de Nanomateriais do Departamento de Física da Universidade Federal de Minas Gerais (UFMG). Nos experimentos será utilizado a bactéria E. coli O157:H7 ATCC 43895 adquirida do banco de culturas da Fiocruz. Os meios microbiológicos usados no estudo serão o Brain Heart Infusion (BHI) caldo, Ágar Bacteriológicos e Ágar Muller Hinton adquiridos da Himedia (Índia).
2.2. Propagação do Fago
O fago para se propagar ele deve infectar uma célula hospedeira. A propagação do fago utiliza solução de ágar base 3,6 g; Tampão SM de 300 mL; solução de Tris HCL (1,18 g); solução de NaCl (0,87 g); solução de MgSO4 (0,3 g); solução de gelatina (1,5 mL). Foram adicionados em microgotas os fagos nas placas contendo ágar base e sobrecamada com uma cultura ativa da bactéria (E.coli). Então incubamos as placas a 30°C por 24h. Em seguida, 10 ml de tampão SM será adicionada nas placas e assim colocadas em um shaker a 17°C por 24h na rotação de 100 rpm. Os fagos foram centrifugados por 20 min a 4°C contendo 4000 g em tubos Falcon com adição de 10% clorofórmio e NaCl 1 mol·L-1. Feito a centrifugação dos fagos, o volume recuperado foi de 25 mL de cada solução de fago.
2.2.2. Titulação do Fago
A solução do fago foi testada para verificação do título (PFU/ml). Com isso, o fago foi diluído em série ( a ) em tampão SM. Foram colocados 500 L da bactéria em sobrecamada vertidas em placas contendo BHI. Após secagem as placas foram demarcadas em quadrantes até diluição . Em seguida, 10 L da diluição do fago foi adicionado em seu respectivo quadrante. Feita a diluição, após a secagem das microgotas, as placas foram incubadas a 37°C por 24h.
2.3. Interação fago-CNT
2.3.1. Avaliação da viabilidade dos fagos após interação com CNT
Para avaliar se houve a interação do fago com o CNT enumerou-se as placas de lise formadas após contato fago-CNT com a bactéria. Para isso, 200 µL de uma cultura pré-ativada de E. coli O157:H7 será espalhada em ágar BHI, deixando-se secar. Será adicionado ao microtubo fago-CNT (de cada tratamento) 1,0 mL de tampão SM e será feita às diluições em série e adicionando 10 µL da alíquota da diluição na placa contendo a bactéria, pela técnica de microgotas. As análises serão realizadas em triplicata para determinar as unidades formadoras de placas por mL da amostra (PFU·mL-1) e, assim, elucidar a taxa de adsorção do fago. As placas serão incubadas a 37 ° C por (18-24h) e as placas de lise formadas serão contadas.
2.4. Montagem do nanosensor
O fago na concentração de PFU· presente no nanotubo (fago-CNT) e a concentração de E. coli O157:H7 de 108 UFC. , serão adicionados, em intervalos de tempo diferentes, em 10 mL de solução tampão SM (50 mmol∙L-1 Tris-HCl [pH 7,5], 0,1 mol∙L-1 NaCl, 8 mmol∙L-1 MgSO4∙7H2O, 0,01 % (m/v) gelatina) que estarão colocado em uma placa de Petri. Antes de ser adicionado na placa, o microtubo contendo fago-CNT será adicionado de 1,0 mL do mesmo tampão e logo após, adicionado na placa. Em seguida, 100 µL da bactéria pré-ativada será adicionada ao tampão contendo o fago-CNT. Proceder-se, então, a leitura da amostra, em função da atuação do fago-CNT, mostrado na forma de diagrama de blocos na Figura 1. Já que a corrente na solução é constante, as variações de tensão sobre ela representam, em última instância, mudanças na sua resistividade oriundas da interação entre as bactérias e o fago-CNT.
Figura 1 – Diagrama em blocos do sistema de medição da resistência elétrica da solução

Figura 2 – Circuito eletrônico do sensor
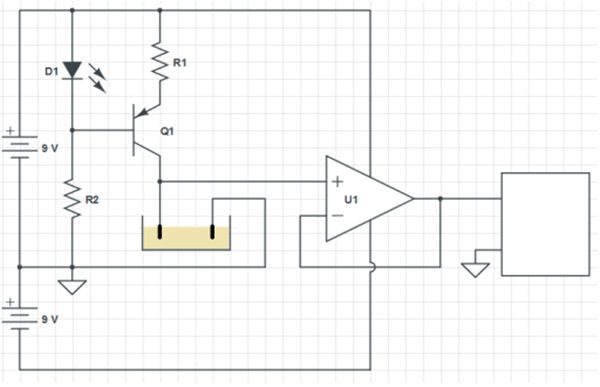
Tabela 2 – Componentes do sensor

Para facilitar o processo de mensuração periódica da tensão aplicada sobre a solução (tampão SM + fago-CNT + bactéria), será utilizada uma plataforma de desenvolvimento de baixo custo em placa única baseada num microcontrolador Atmel AVR, com hardware e software abertos. O modelo empregado foi o MEGA da família Arduino, mostrado na Figura 3.
Figura 3 – Arduíno MEGA

O firmware (código-fonte) do microcontrolador dessa plataforma será escrito para fazer as leituras da tensão com intervalos de 20 s, enviando-as então para um computador, onde elas foram salvas em um arquivo para posterior análise.
2.5 Protótipo do nanosensor
Ao analisar o resultado obtido anteriormente e sua repetibilidade, será proposto um protótipo de nanosensor via desenho técnico de um circuito eletrônico, aquisição de componentes eletrônicos, impressão em 3D da carcaça do sensor e teste desse protótipo com E. coli.
RESULTADOS E DISCUSSÃO
A solução que continha o fago-CNT e a bactéria passou por vários testes de leitura de voltagem para determinar o circuito a ser construído. O protótipo final foi então projetado e montado. Uma vez montado o protótipo do nanossensor para leitura da solução (tampão SM + fago-CNT + bactéria), a leitura do meio foi estabilizada. Aos 4 minutos foi adicionado o fago-CNT e após 3 minutos foi incluída a bactéria pré-ativada ( UFC.mL-1). A leitura foi realizada durante uma 1h e 30min.
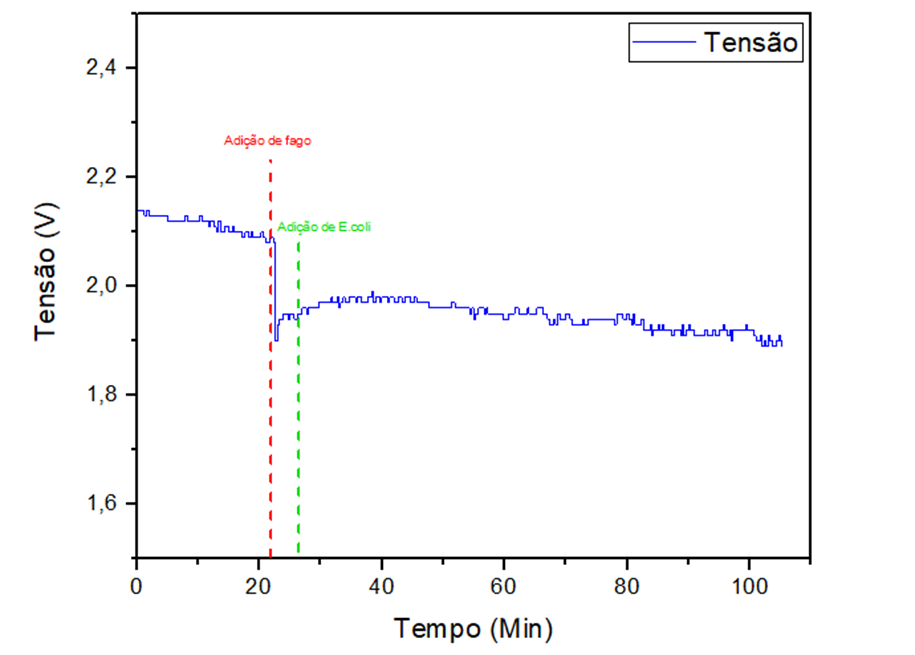
Figura 4 – gráfico da tensão versus tempo da amostra contendo fago-CNT mais a bactéria E. coli O157:H7
Após aproximadamente 23 min do contato inicial entre a bactéria e o fago, a voltagem apresentou um pico. Este pico resultou da quebra das células bacterianas, levando à lise celular e consequente liberação de íons do interior da célula para o meio circundante. A liberação de íons alterou a resistividade do meio. A cinética da investigação do fago UFV-AREG1 revelou que leva cerca de 40-50 minutos para o fago infectar a célula e causar a morte celular, como o teste do fago chegou aproximadamente 23min, serão realizados mais testes com concertações diferentes de fago-CNT e bactéria E.coli O157:H7. Após esse período, ocorre o burst-size, que provoca a liberação de cerca de 18 partículas virais por célula infectada. A parede celular da bactéria contém receptores específicos que o fago deve reconhecer para injetar seu material genético no hospedeiro. Ocasionalmente, modificações genéticas no capsídeo viral são necessárias para o desenvolvimento de certos sensores, pois permitem a imobilização do fago na superfície do sensor, o que é crucial para a detecção eficaz do patógeno.
Um nanobiossensor capaz de medir a voltagem de maneira econômica e direta foi criado usando a cinética do fago como base. A quantidade de tempo que os fagos gastam infectando e lisando células determina a taxa de detecção bacteriana pelo sensor, com períodos mais curtos resultando em detecção mais rápida. Além disso, este nanobiossensor pode determinar o tamanho da explosão do fago utilizado, tornando-se um recurso valioso no estudo da cinética. Nanotubos de carbono de paredes múltiplas foram utilizados para amplificação de sinal e imobilização de fagos devido à sua maior área de superfície, o que permite a produção de dispositivos multifuncionais com uma pequena área de seção transversal. Este aumento na área de superfície também permite um aumento no sinal pretendido usando a molécula de interesse. Com a utilização de vários fagos, o método aqui estabelecido tem potencial para construir nanobiossensores que podem detectar hospedeiros específicos com base em sua identificação.
CONCLUSÕES
A eficácia da imobilização do bacteriófago UFV-AREG1 em CNT foi confirmada pelos resultados da estabilidade e viabilidade do vírus no nanotubo. O experimento foi projetado e conduzido de forma a produzir uma análise estatística que não revelou nenhuma perda viral significativa na carga que foi adsorvida no nanotubo. A atividade antimicrobiana no material preparado foi confirmada pelo teste de disco-difusão. O CNT foi capaz de detectar E. coli O157:H7 e o tempo de explosão do vírus quando o fago imobilizado foi conectado ao circuito eletrônico do nanobiossensor. Este mecanismo pode ser utilizado com outros fagos imobilizados no CNT para detectar o hospedeiro desejado ou para determinar o tamanho da explosão viral. Testes futuros serão realizados para otimizar o nanobiossensor para uso comercial na indústria de análise de alimentos.
REFERÊNCIAS
- Lopez-Velasco G, Tomas-Callejas A, Sbodio AO, Pham X, Wei P, Diribsa D, et al. Factors affecting cell population density during enrichment and subsequent molecular detection of Salmonella enterica and Escherichia coli O157:H7 on lettuce contaminated during field production. Food Control. 2015 Aug;54:165–75.
- Scallan E, Griffin PM, Angulo FJ, Tauxe R V., Hoekstra RM. Foodborne Illness Acquired in the United States—Unspecified Agents. Emerg Infect Dis. 2011 Jan;17(1):16–22.
- Tadese ND, Gebremedhi EZ, Moges F, Borana BM, Marami LM, Sarba EJ, et al. Occurrence and Antibiogram of Escherichia coli O157 : H7 in Raw Beef and Hygienic Practices in Abattoir and Retailer Shops in Ambo Town, Ethiopia. Vet Med Int. 2021 Apr 1;2021:1–12.
- Trigo IA, Yada MM, Lourençano L da S, Lima YK de. USO DE TECNOLOGIA NA RASTREABILIDADE DO REBANHO DE CORTE. Revista Interface Tecnológica. 2018 Dec 30;15(2):381–91.
- Rani A, Ravindran VB, Surapaneni A, Mantri N, Ball AS. Review: Trends in point-of-care diagnosis for Escherichia coli O157:H7 in food and water. Int J Food Microbiol. 2021 Jul;349:109233.
- HOORFAR J. Rapid detection, characterization, and enumeration of foodborne pathogens. APMIS. 2011 Nov;119:1–24.
- Wang Y, Ye Z, Ying Y. New Trends in Impedimetric Biosensors for the Detection of Foodborne Pathogenic Bacteria. Sensors. 2012 Mar 12;12(3):3449–71.
- Hungaro HM, Mendonça RCS, Gouvêa DM, Vanetti MCD, Pinto CL de O. Use of bacteriophages to reduce Salmonella in chicken skin in comparison with chemical agents. Food Research International. 2013 Jun;52(1):75–81.
- Horgan M, O’Sullivan O, Coffey A, Fitzgerald GF, van Sinderen D, McAuliffe O, et al. Genome analysis of the Clostridium difficile phage ΦCD6356, a temperate phage of the Siphoviridae family. Gene. 2010 Aug;462(1–2):34–43.
- Cooper IR, Illsley M, Korobeinyk A V., Whitby RLD. Bacteriophage-nanocomposites: An easy and reproducible method for the construction, handling, storage and transport of conjugates for deployment of bacteriophages active against Pseudomonas aeruginosa. J Microbiol Methods. 2015 Apr;111:111–8.
- Mahony J, McAuliffe O, Ross RP, van Sinderen D. Bacteriophages as biocontrol agents of food pathogens. Curr Opin Biotechnol. 2011 Apr;22(2):157–63.
- Iijima S, Ajayan PM, Ichihashi T. Growth model for carbon nanotubes. Phys Rev Lett. 1992 Nov 23;69(21):3100–3.
- Katz E, Willner I. Biomolecule-Functionalized Carbon Nanotubes: Applications in Nanobioelectronics. ChemPhysChem. 2004 Aug 20;5(8):1084–104.
- Lopez MES, Batalha LS, Vidigal PMP, Albino LAA, Boggione DMG, Gontijo MTP, et al. Genome Sequence of the Enterohemorrhagic Escherichia coli Bacteriophage UFV-AREG1. Genome Announc. 2016 Oct 27;4(5).